根据代表性序列预测OTU/ASV生活史策略——寡营养型or富营养型
定期体检,根据自身需求调整营养搭配策略。 #生活技巧# #健康生活方式# #营养搭配策略#
一、原理介绍
丰富的资源会促进细菌的生长,特别是富营养型细菌的生长。快速的生长需要大量的核糖体,富营养性细菌会持有更多的核糖体RNA操纵子(number of ribosomal RNA operons, rrn)。因此我们可以根据核糖体RNA操纵子的数目来对寡营养型和富营养型细菌进行区分,而且核糖体RNA操纵子的数目在16srRNA序列上保守,因此我们可以通过分类信息对核糖体RNA操纵子数进行预测。
二、实现过程
1.对代表性序列利用RDP分类器进行分类,获得RDP注释的OTU:
java -Xmx1g -jar ./dist/classifier.jar classify -c 0.5 -o classified.txt yourpath/rep_otus_seqs.fasta
如果命令行运行困难可使用RDP的网站进行分类注释:http://rdp.cme.msu.edu/classifier/classifier.jsp
该RDP分类器的命令行工具可在如下网址下载:https://sourceforge.net/projects/rdp-classifier/files/latest/downloa
代表性序列长这样:
输出结果长这样:
将获得的注释数据与rrnDB数据库按从genus到domain的顺序进行比较,配对上的核糖体RNA操纵子数目(number of ribosomal RNA operons, rrn)的平均值即为该OTU的rrn。
rrnDB数据库长这样:
比对的R代码:
classified <- readLines("~/rnn_predictor/classified.txt")
rrn <- read.delim("~/rnn_predictor/rrnDB-5.7_pantaxa_stats_RDP.tsv", header=TRUE)
rrn2 <- data.frame(rank = rrn$rank,name = rrn$name,mean = rrn$mean)
rm(rrn)
taxid = rep(0,length(classified))
rnn_copy = rep(0,length(classified))
res = data.frame(taxid,rnn_copy)
for (i in 1:length(classified)) {
str <- strsplit(classified[i],"\t")[[1]]
str2 <- str[which(str %in% c("genus","family","order","class","phylum","domain"))-1]
res$taxid[i] = str[1]
for (j in (0:length(str2))){
if (j == length(str2)){
res$rnn_copy[i] = "NA"
break
}
if(str2[length(str2)-j] %in% rrn2$name){
print(i)
res[i,2] = rrn2[rrn2$name == str2[length(str2)-j],3][1]
break
}
}
}
write.table(res,"~/Desktop/rnn_predict.txt",sep="\t",row.names=FALSE,col.names=TRUE,quote=FALSE)
获得的结果长这样:
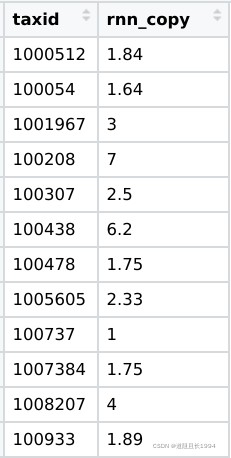
这样我们就获得了每个OTU的核糖体RNA操纵子数目(number of ribosomal RNA operons, rrn),富营养型的细菌往往有高的16srRNA基因的携带数,寡营养型细菌的16srRNA基因的携带数则较低,因此我们可以根据我们预测的结果推断该OTU的生活史策略。
修改:上面的代码写于一个月前,现在来看觉得不符合R语言的编程习惯。上述代码使用了过多的for循环,特别是嵌套的for循环,这在R语言中是不提倡的,因为当循环过多时会严重制约运行速度,因此上述代码不适用于OTU数目较多(>5000)的情况。因此对上述的R代码进行了修改,但依然保持原来的代码作为提醒,写代码时要时常问自己这和问题:代码的计算量随数据量的增加是指数增加还是线性增加?
修改后的代码如下:
classified <- readLines("./classified.txt")
rrn <- read.delim("../rrnDB-5.7_pantaxa_stats_RDP.tsv", header=TRUE)
rrn2 <- data.frame(rank = rrn$rank,name = rrn$name,mean = rrn$mean)
rm(rrn)
strwhich <- function(xstr) {
str <- strsplit(xstr,"\t")[[1]]
return (c(str[1],str[which(str %in% c("genus","family","order","class","phylum","domain"))-1]))
}
a <- lapply(classified,strwhich)
maptax2rnn <- function(xstr){
n <- tail(which(unlist(lapply(xstr,function(x) x%in%rrn2$name))),1)
return(c(xstr[1],rrn2$mean[which(rrn2$name == xstr[n])[1]]))
}
b <- lapply(a, maptax2rnn)
res <- matrix(unlist(b),nrow=length(b),ncol=length(b[[1]]),
byrow=TRUE)
colnames(res)<-c("OTUID","rnn_copies")
write.table(res,"./rnn_predict.txt",sep="\t",row.names=FALSE,col.names=TRUE,quote=FALSE)
可通过以下资源重复上述过程:
根据代表性序列预测OTU/ASV生活史策略——寡营养型or富营养型-专业指导文档类资源-CSDN下载丰富的资源会促进细菌的生长,特别是富营养型细菌的生长。快速的生长需要大量的核糖体,富营养性细菌会持更多下载资源、学习资料请访问CSDN下载频道.https://download.csdn.net/download/weixin_43367441/80426535
更多R语言分析微生物生态学的资源可参考如下链接:
微生物共现网络与群落构建分析https://mianbaoduo.com/o/works/240342
参考文献:
1. Exploiting rRNA operon copy number to investigate bacterial reproductive strategies
2. Nutrient supply controls the linkage between species abundance and ecological interactions in marine bacterial communities
觉得博主写地不错的话,可以买箱苹果,支持博主。
网址:根据代表性序列预测OTU/ASV生活史策略——寡营养型or富营养型 https://www.yuejiaxmz.com/news/view/834304
相关内容
iMeta|中科院地球环境研究所王云强组解析了草地土壤微生物生活史策略营养代谢病预防策略
科学网—科研丨ISME:干旱土壤生态系统中微生物群落的生活史策略
表观基因组:基于基因数据的个性化营养和生活方式干预
耐力运动表现的膳食营养策略分析
综述
健康饮食新风尚:预防慢性病的营养策略
中国营养学会发布:《新时代慢性病防治食养食疗策略和行动》白皮书
绿色生活:环保型房地产的营销策略.docx
心肌肥厚的营养管理:饮食与营养补充的策略.pptx

